Egy gyors karakterizálásra alkalmas DNS vonalkód bevezetéséről határoztak növénybiológusok egy most zajló konferencián. A vonalkódot két olyan gén képezi amely minden növényben megtalálható, de fajonként némileg eltér egymástól, így egyedi jelölőként funkcionál. A módszerrel lehetővé válik természetvédelmi területek kijelölésénél a jelen lévő védett növények kimutatása, vagy vitatott hovatartozású növények gyors azonosítása.
 2009 november 10-én sikerült a Mexikóvárosban megtartott 3. International Barcode of Life konferencia keretein belül megállapodni arról, hogy innentől kezdve minden szárazföldi növényt DNS vonalkód alapján azonosítsanak. A döntés értelmében 150 millió dollárból készül el 5 év alatt egy 5 millió vonalkódot tartalmazó könyvtár, melynek segítségével nagyjából félmillió növényfaj lesz azonosítható. "Ez a legnagyobb biodiverzitás genomikai program amire valaha is vállalkoztak" mondta el Paul Hebert a University of Guelph in Ontario munkatársa aki még 2003-ban rukkolt elő a DNS vonalkódozás ötletével.
2009 november 10-én sikerült a Mexikóvárosban megtartott 3. International Barcode of Life konferencia keretein belül megállapodni arról, hogy innentől kezdve minden szárazföldi növényt DNS vonalkód alapján azonosítsanak. A döntés értelmében 150 millió dollárból készül el 5 év alatt egy 5 millió vonalkódot tartalmazó könyvtár, melynek segítségével nagyjából félmillió növényfaj lesz azonosítható. "Ez a legnagyobb biodiverzitás genomikai program amire valaha is vállalkoztak" mondta el Paul Hebert a University of Guelph in Ontario munkatársa aki még 2003-ban rukkolt elő a DNS vonalkódozás ötletével.
A módszer végre jelentősen lerövidítheti azt a procedúrát amit egy-egy növény meghatározása jelentett. Az időigényes morfológián (azaz a növény külső megjelenésén) alapuló meghatározás ugyanis erősen szubjektív és nem utolsósorban igencsak időigényes. Egy vonalkód leolvasás viszont ehhez képest rövid idő alatt megvan, és igen megbízható eredményt is ad. Az előzetes becslések szerint az esetek 99%-ában alkalmazható lesz a módszer, és az elsődleges tesztek alapján 72%-os valószínüséggel állapítható meg vele a helyes faj. Ez elsőre nem feltétlenül tűnik túl meggyőzőnek, de ha hozzávesszük, hogy a módszer 100%-os biztonsággal helyezi a megfelelő fajcsoportba a meghatározandó növényt, akkor máris látható a módszer haszna.
Az eljárás lényege, hogy két olyan gént elemeznek a tudósok a vizsgálandó növény DNS állományában, ami minden növényben jelen van, de fajonként eltérő. A kapott génszekvenciát (azaz a kapott DNS darabot) összevetik a vonalkód adatbázissal, az egyező kód alapján pedig azonosítják a növényt. A módszernél kulcsfontosságú, hogy pontosan mely DNS darabokat használnak a kódolásra, ez ugyanis döntően befolyásolja a rendszer hatékonyságát. Hosszas előkészítés után végül az rbcL és a matK génekre esett a választás.
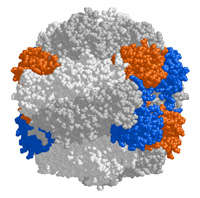 Az rbcL gén egy létfontosságú enzimet kódol (teljes nevén a ribulóz 1,5-biszfoszfát karboxiláz oxigenázt) ami elengedhetetlen ahhoz, hogy a növények szén-dioxidból saját szerves anyagaikat felépíthessék. Az enzim olyannyira fontos, hogy minden zöld levélben termelődik, és becslések szerint az egyik legnagyobb mennyiségben jelen lévő fehérje a Földön. Az általában rubisco-nak rövidített enzim két külön alegységből áll, az rbcL a "large", azaz a nagyobbik alegységet kódolja. A gén univerzálissága miatt úgynevezett molekuláris óraként is funkcionál, ugyanis közeli rokon fajok között kisebb eltéréseket mutat, mint távoliaknál. Két növényfaj rbcL-ének összehasonlításával emiatt felmérhető, hogy nagyjából mennyi ideje válhatott szét a két csoport egymástól.
Az rbcL gén egy létfontosságú enzimet kódol (teljes nevén a ribulóz 1,5-biszfoszfát karboxiláz oxigenázt) ami elengedhetetlen ahhoz, hogy a növények szén-dioxidból saját szerves anyagaikat felépíthessék. Az enzim olyannyira fontos, hogy minden zöld levélben termelődik, és becslések szerint az egyik legnagyobb mennyiségben jelen lévő fehérje a Földön. Az általában rubisco-nak rövidített enzim két külön alegységből áll, az rbcL a "large", azaz a nagyobbik alegységet kódolja. A gén univerzálissága miatt úgynevezett molekuláris óraként is funkcionál, ugyanis közeli rokon fajok között kisebb eltéréseket mutat, mint távoliaknál. Két növényfaj rbcL-ének összehasonlításával emiatt felmérhető, hogy nagyjából mennyi ideje válhatott szét a két csoport egymástól.
A szintén használt matK egy kevésbé ismert gén, feltehetőleg más RNS molekulák érésében játszik szerepet. Jelen szempontból viszont azért fontos, mert ez is univerzális növényi fehérje, és már korábbi vizsgálatokban is sikerrel vizsgázott növényhatározás terén.
Ugyan a két gént már eddig is használták növényhatározásra, és többen is ezeket jelölték a vonalkód alapjának, a végleges döntés mégis csak most született meg. Az előkészítő bizottság ugyanis csak nehezen jutott konszenzusra, egyes tagok egy három génes megoldást javasoltak, mások a két gén mellé speciális "biztonsági régiókat" láttak szükségesnek. A döntés még így sem végleges, a mostani bejelentés után még ugyanis 18 hónap múlva felülvizsgálják a vonalkód helyességét.
"Mindenki erre a döntésre várt már időtlen idők óta" mondta el David Schindel a Barcode of Life konzorcium főtitkára. A most lefektetett alapokon ugyanis végre megindulhat a vonalkód könyvtár építése, ami remélhetőleg néhány éven belül már rutinszerűen alkalmazható lesz a szárazföldi növények meghatározására.
Forrás:
The Great Beyond








Utolsó hozzászólások